“For mechanistic studies of DNA repair”
Per l’anno 2015 il premio Nobel per la Chimica è stato attribuito a tre ricercatori che hanno dato un contributo fondamentale alla scoperta e alla comprensione di un fenomeno di importanza primaria nel corretto funzionamento e nella stessa sopravvivenza di tutti gli organismi, inclusa naturalmente la nostra specie. Il premio è stato loro attribuito «for mechanistic studies of DNA repair», come recita la motivazione ufficiale, che si potrebbe tradurre: «per i loro studi sui meccanismi della riparazione del DNA».
È oggi nozione comune che l’acido deossiribonucleico (DNA) è il depositario dell’informazione genetica, che ultimamente si attua nella sintesi di un repertorio di proteine. Queste, a loro volta, sono molecole che attuano virtualmente tutte le funzioni di un organismo. In particolare, molti tratti di DNA (ma non tutti) codificano proteine, e i tratti codificanti sono denominati geni.
L’informazione genetica è codificata nel DNA in base ad un «alfabeto» di 4 lettere. In termini molecolari, le lettere sono le basi azotate adenina, timina, guanina e citosina, simbolizzate rispettivamente con le lettere A, T, G e C (si veda le Figure 1 che seguono), che sono assemblate in una sequenza lineare su uno scheletro la cui struttura è costante lungo tutta la lunghezza, ed è composta da un particolare zucchero, il deossiribosio, e da fosfato.
Il DNA è costituito da una sequenza di nucleotidi, che ne rappresentano le unità base, e ciascun nucleotide consiste di un deossiribosio legato a un fosfato e a una delle quattro basi (come rappresentato nelle Figure 1 che seguono).
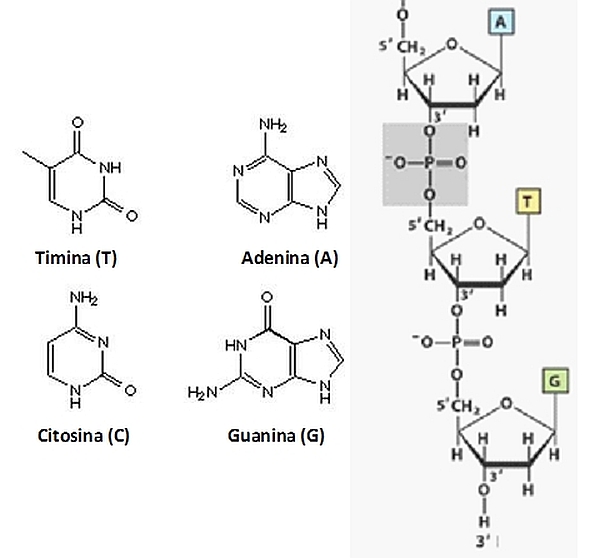 Figura 1A. Gli aspetti strutturali essenziali del DNA: le quattro basi azotate come tali e all’interno dei relativi nucleotidi, legati tra loro a formare una molecola di DNA.
Figura 1A. Gli aspetti strutturali essenziali del DNA: le quattro basi azotate come tali e all’interno dei relativi nucleotidi, legati tra loro a formare una molecola di DNA.
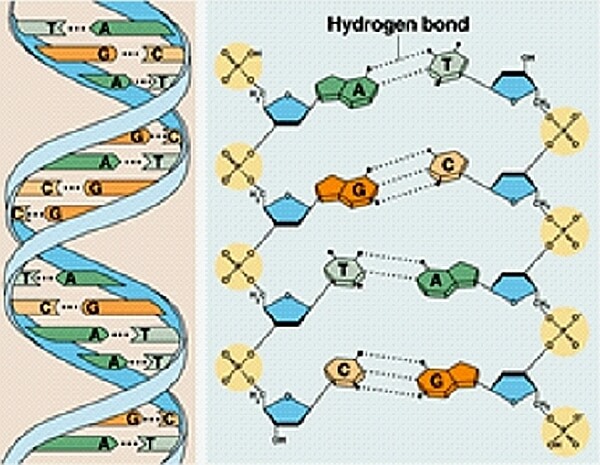 Figura 1B. Gli aspetti strutturali essenziali del DNA: due diverse raffigurazioni di una molecola di DNA, completa dei due filamenti appaiati
Figura 1B. Gli aspetti strutturali essenziali del DNA: due diverse raffigurazioni di una molecola di DNA, completa dei due filamenti appaiati
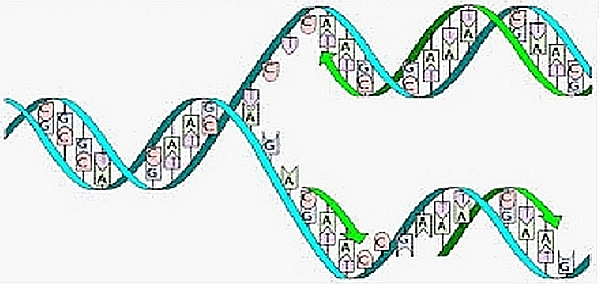 Figura 1C. Gli aspetti strutturali essenziali del DNA: a rappresentazione schematica della duplicazione di una molecola a dare due molecole figlie identiche
Figura 1C. Gli aspetti strutturali essenziali del DNA: a rappresentazione schematica della duplicazione di una molecola a dare due molecole figlie identiche
Un’altra importante proprietà strutturale di questa macromolecola è che essa è formata di norma da due filamenti appaiati (come illustrato nella stessa figura). I due filamenti si ravvolgono l’uno sull’altro elicoidalmente, generando la ben nota doppia elica.
Oltre a ciò, un altro aspetto di un enorme rilievo è che, mediante interazioni chimiche dette legami idrogeno, e grazie a altre proprietà strutturali, ciascuna base di un filamento si appaia a una base del filamento prospiciente secondo regole semplici e ben definite: in sostanza, salvo errori, l’adenina si può solo appaiare alla timina e la guanina alla citosina.
Le regole di appaiamento implicano che ciascuno dei due filamenti di DNA contengano la stessa informazione, sia pur codificata secondo modalità complementari. Questa importantissima caratteristica ha suggerito, già all’atto della definizione della struttura da parte di James D. Watson (1928-..) e Francis Crick (1916-2004), gli aspetti essenziali della modalità con cui le molecole di DNA si duplicano. Tale modalità comporta in linea teorica la perfetta conservazione nelle molecole figlie della sequenza in basi e quindi della informazione contenuta nella molecola madre (si veda le Figure 1 precedenti).
Date queste premesse, non risulta difficile cogliere l’importanza del mantenimento della integrità strutturale del DNA, come pure della sua corretta sequenza in basi. Modificazioni che compromettessero severamente queste caratteristiche potrebbero infatti portare a importanti difetti nel «funzionamento» dell’organismo. A tali modificazioni è infatti associato un rischio di malattie genetiche, trasmesse attraverso la linea germinale, e anche di malattie tumorali, che invece si sviluppano a seguito di alterazioni del genoma nelle cellule somatiche.
In effetti tali fenomeni sono relativamente rari, e purtuttavia il DNA non è una struttura immutabile: è difatti nozione comune che in esso si possono accumulare modificazioni, generalmente denominate mutazioni, che includono ampie variazioni in sequenza, come inserzioni, delezioni o duplicazioni di tratti interi; oppure, nel caso più semplice si tratta della sostituzione di una determinata base con un’altra. Anzi, tali fenomeni sono fondamentali nell’evoluzione biologica, che implica necessariamente una modificazione del patrimonio genetico nel tempo, e che porta alla transizione da una specie a un’altra. Ciò nondimeno, nel breve periodo il genoma di un organismo (vale a dire, l’insieme del DNA presente in ciascuna sua cellula) resta sostanzialmente costante.
Fino agli inizi degli anni Sessanta del secolo scorso, si riteneva che il DNA fosse intrinsecamente stabile, cioè poco soggetto a subire modificazioni chimiche indipendentemente dall’esistenza di sistemi di protezione della sua integrità. In realtà, successive investigazioni dimostrarono che ciò non era affatto vero.
Un contributo fondamentale in questo senso fu offerto dallo svedese Tomas Lindhal, uno dei tre vincitori del premio Nobel di quest’anno. Dopo aver conseguito il dottorato all’Istituto Karolinska nel 1967, nei suoi studi successivi presso la stessa istituzione poté dimostrare, nel corso di investigazioni sul virus di Epstein Barr (responsabile della mononucleosi), che in cellule umane il DNA subiva migliaia di modificazioni di diverso tipo addirittura nel corso di una singola giornata. La causa di questa instabilità è legata a una molteplicità di fattori, alcuni interni alla cellula come la produzione di radicali liberi (un prodotto inevitabile del metabolismo ossidativo), estremamente reattivi; altri esterni, quali per esempio i raggi ultravioletti (UV).
A fronte di questo stava il fatto della sua sostanziale stabilità. Da questa osservazione prese le mosse la sua successiva ricerca, svolta ancora al Karolinska, e successivamente all’Università di Göteborg e quindi all’Imperial Cancer Research Fund di Londra, che lo portò a scoprire e caratterizzare uno dei sistemi di controllo dell’integrità del DNA, denominato in inglese base excision repair (riparazione per escissione di base).
Tale meccanismo può rimediare a eventi che portino la sostituzione di una base corretta con una «errata»: in particolare la citosina, una delle quattro basi che nella doppia elica si appaia normalmente con la guanina (si veda le Figure 1 precedenti), perde spesso e spontaneamente un gruppo amminico per deamminazione generando uracile, una base molto simile alla timina e che per questo è in grado di appaiarsi con adenina.
Il risultato di questo fenomeno è che nel corso della duplicazione del DNA, nella posizione in cui originariamente esisteva la coppia di basi citosina-guanina, viene a trovarsi la coppia adenina-timina. Ebbene, il meccanismo di base excision repair porta alla rimozione dell’uracile e alla reintroduzione della base corretta, vale a dire la citosina (si veda la Figura 2).
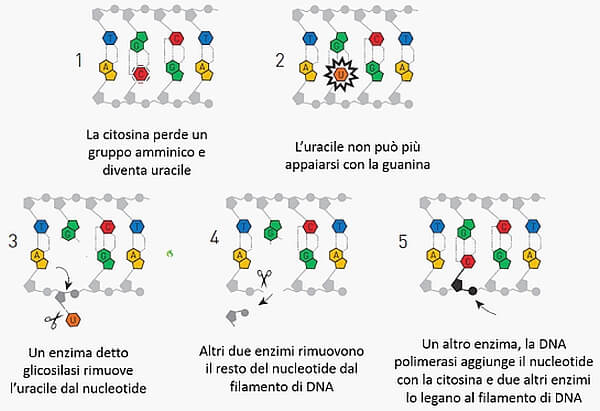 Figura 2. Il meccanismo del base excision repair
Figura 2. Il meccanismo del base excision repair
In realtà, le scoperte di Lindhal erano solo una parte della storia: altri meccanismi non meno importanti sono stati scoperti successivamente. Aziz Sancar, un medico turco, decise di dedicarsi a studi di biochimica e per questo si trasferì a Dallas, all’Università del Texas, a metà degli anni ‘70 del secolo scorso.
Il suo interesse scientifico si focalizzò sui meccanismi di riparazione dei danni indotti da raggi UV sul DNA, che consistono principalmente nella dimerizzazione di basi, soprattutto le timine. Ciò significa, in altre parole, che due basi consecutive sullo stesso filamento formano tra loro legami covalenti e quindi stabili. Ciò modifica la struttura complessiva del DNA, e può impedire o rallentare la sua replicazione, ma anche introdurre delle mutazioni durante la replicazione.
È degno di nota che questa alterazione del DNA può essere causata non solo da raggi UV, ma anche da sostanze carcinogeniche come alcuni idrocarburi presenti nel fumo di sigaretta.
In Texas Sancar investigò un sistema di riparazione attivo nei batteri ma non nell’uomo; ma le scoperte che gli valsero la notorietà vennero in seguito, dopo che egli si trasferì all’Università di Yale, e successivamente a quella della North Carolina. Soprattutto negli anni Ottanta, egli chiarì il funzionamento dell’unico sistema di riparazione di questi danni esistente nell’uomo e negli altri mammiferi placentali, denominato nucleotide excision repair (riparazione per escissione di nucleotidi).
Tale sistema ha un meccanismo di funzionamento molto diverso da quello del base excision repair: gli enzimi che lo attuano rimuovono infatti un tratto di 12 nucleotidi attorno al punto di dimerizzazione e lo sostituiscono con un tratto nuovo e senza difetti (si veda la Figura 3).
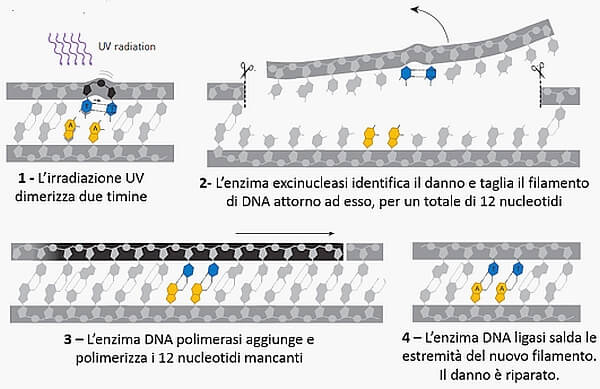 Figura 3. Il meccanismo del nucleotide excision repair
Figura 3. Il meccanismo del nucleotide excision repair
Il terzo vincitore del premio Nobel è Paul Modrich, un americano nato nel New Messico il quale iniziò la sua carriera alla Università di Stanford, dove nel 1973 conseguì il dottorato, proseguì i suoi studi per un breve periodo ad Harvard, e successivamente, nel 1976, si trasferì alla Duke University dove ancora oggi è professore.
Fin dall’inizio della sua carriera, Modrich si interessò agli enzimi capaci di modificare il DNA e replicarlo. Lo spunto all’origine della sua ricerca, e che infine lo portò alle scoperte che gli hanno valso il premio Nobel, gli venne dalla collaborazione con un altro prominente scienziato, Matthew Meselson, quando entrambi erano ad Harvard. Meselson si occupava della metilazione del DNA. Questa è una modificazione chimica consistente nell’aggiunta di un piccolo gruppo, il metile, a determinate basi.
La ricerca di Meselson si proponeva di valutare se questa modificazione fungesse da segnale di indirizzamento per certi enzimi capaci di tagliare in modo molto selettivo il DNA in ben precise posizioni (i cosiddetti enzimi di restrizione). A questo scopo aveva costruito un virus, capace di infettare dei batteri, che nel suo DNA possedeva parecchi appaiamenti scorretti di basi (in inglese: mismatch). Tuttavia quando questi virus infettavano i rispettivi batteri, tali mismatch del DNA virale iniettato venivano eliminati.
Ciò portò Meselson a speculare che nei batteri potesse esistere un meccanismo di riparazione capace di correggere questi appaiamenti erronei, che talvolta si verificano quando il DNA si replica. Sulla base sei suoi risultati, egli ipotizzò inoltre che i gruppi metile fossero aggiunti al filamento portante la sequenza giusta e che quindi quello da correggere fosse quello complementare.
Queste iniziali scoperte stimolarono Modrich a intraprendere un lavoro di caratterizzazione sistematica degli enzimi che compongono il sistema di riparazione, denominato DNA mismatch repair. Verso la fine degli anni Ottanta egli aveva largamente delucidato il meccanismo di questo complesso sistema nell’uomo (si veda la Figura 4).
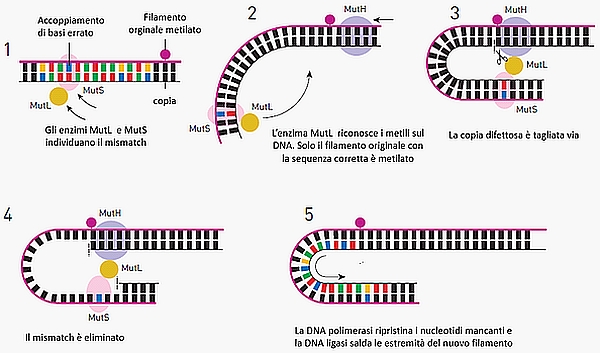 Figura 4. Il meccanismo del DNA mismatch repair
Figura 4. Il meccanismo del DNA mismatch repair
Quali le implicazioni di queste scoperte? Oggi sappiamo che tali meccanismi sono così efficienti da correggere almeno il 99,9% di tutti gli errori che vengono introdotti nella sequenza del DNA.
In particolare il mismatch repair corregge soprattutto quelli introdotti durante la sua replicazione. Il nostro genoma degenererebbe rapidamente in assenza di tutti questi meccanismi. Sappiamo anche che loro difetti sono responsabili di diverse malattie: in particolare, in certe forme di cancro almeno uno di questi sistemi di riparazione è difettoso.
Non è difficile immaginare che queste scoperte offrano delle potenziali ricadute applicative di notevole importanza: per esempio, sono allo studio agenti antitumorali in grado di inattivare selettivamente i sistemi di riparazione del DNA nelle cellule cancerose. Se questo obiettivo fosse conseguito, esse andrebbero incontro rapidamente a morte.
Paolo Tortora
(Ordinario di Biochimica, presso il Dipartimento di Biotecnologie e Bioscienze dell’Università degli Studi di Milano Bicocca)
© Pubblicato sul n° 59 di Emmeciquadro
